Information for the public
COVID-19: Drei Kontakte mit dem Spike-Protein für eine breite Immunität
Seit Beginn der COVID-19-Pandemie verändert sich das Coronavirus SARS-CoV-2 immer weiter. Neue besorgniserregenden Varianten (variants of concern, VOC) breiten sich rasant aus. Omikron, das hochansteckend ist und sich der Immunantwort teilweise entziehen kann, ist in den meisten Ländern zur dominierenden Variante geworden.
Die Varianten Alpha und Delta werden leichter übertragen als das ursprüngliche SARS-CoV-2. Andere Varianten umgehen die Antwort des Immunsystems gegen das ursprüngliche Virus – zumindest zum Teil. Das gilt für Beta, Gamma und Delta. Auf die Omikron-Variante trifft beides zu. Omikron weist umfangreiche Mutationen im Spike-Protein auf, was seine außerordentlich schnelle Verbreitung und seine Immunflucht erklärt: eine Herausforderung für Ärztinnen und Ärzte, denn als Frage bleibt, wie es gelingt, Menschen bestmöglich gegen Infektionen mit dieser VOC zu schützen.
Antworten hat ein Team um Prof. Dr. Ulrike Protzer (Institut für Virologie der Technischen Universität München und Helmholtz Munich), Prof. Dr. Percy A. Knolle (Klinikum rechts der Isar der TUM) und Prof. Dr. Oliver T. Keppler (Max von Pettenkofer-Institut und Genzentrum der LMU) gefunden. Wie sie in Nature Medicine berichten, sind insgesamt drei Kontakte zum Spike-Protein als viralem Antigen notwendig, damit neutralisierende Antikörper nicht nur in ausreichender Menge, sondern auch in hoher Qualität gebildet werden. Solche qualitativ hochwertigen Antikörper binden das Virus stärker und können dadurch auch die Omikron-Variante bekämpfen. Das gilt sowohl für dreifach Geimpfte als auch für Genesene nach zwei Impfungen und zweifach Geimpfte nach einer Durchbruchsinfektion.
Längsschnittstudie mit 171 Teilnehmenden
Für die Studie wurden seit Beginn der Pandemie freiwillige Teilnehmer aus dem Kreis der Mitarbeitenden am Klinikum rechts der Isar der TUM rekrutiert und regelmäßig untersucht. Hierbei wurde eine Gruppe identifiziert, die sich in der ersten Pandemie-Welle im Frühjahr 2020 mit SARS-CoV-2 infiziert hatte, und eine zweite Gruppe, die sich nicht infiziert hatte. Später wurden beiden Gruppen Impfungen mit dem mRNA-basierten COVID-19-Impfstoff von BioNTech/Pfizer angeboten, und sie wurden knapp zwei Jahre lang nachverfolgt. Die Kohorte umfasste 98 Genesene und 73 Personen ohne vorherige Infektion. Beide Gruppen waren hinsichtlich ihres Geschlechts, Alters, hinsichtlich der Arbeitsbedingungen und hinsichtlich weiterer Risikofaktoren vergleichbar.
„Diese Längsschnitt-Untersuchung ist besonders spannend, weil man die gleichen Menschen über einen langen Zeitraum verfolgt und ihre T-Zell Immunität regelmäßig untersucht“, sagt Prof. Knolle, und weist auf eine dazu gerade in Nature Communications erschiene Studie des Teams hin. In der aktuellen Studie haben die Forschenden im Blut der Probandinnen und Probanden mehrere Parameter bestimmt: die Menge der Antikörper (IgG), die Stärke der Bindung zwischen Virus-Protein und Antikörper sowie die Fähigkeit von Antikörpern, SARS-CoV-2 Varianten in Zellkultur zu neutralisieren. Die beiden letzteren sind besonders wichtig, um das Ausmaß der schützenden Immunität abzuschätzen. Die Studie zeigte auf, dass die Fähigkeit des Immunsystems, das Virus zu neutralisieren, nur teilweise mit dem Antikörper-Titer korreliert. Vielmehr ist es entscheidend, wie effektiv diese Antikörper an das Virus binden und es so inaktivieren.
Wie aus seiner Struktur vorhergesagt zeigte Omikron im Vergleich zu anderen Varianten die am stärksten ausgeprägte Immunflucht gegenüber neutralisierenden Antikörpern. „Hier braucht man deutlich mehr und bessere Antikörper, um das Virus zu neutralisieren“, kommentiert Prof. Keppler. Die Forscher hatten für die Untersuchungen einen neuen Test entwickelt, mit dem man in hohem Durchsatz viele Serumproben und verschiedene Varianten des Virus in einem Hochsicherheitslabor innerhalb weniger Tage untersuchen kann. Und Prof. Protzer ergänzt: „Eine neue Erkenntnis aus unserer Studie ist, dass Menschen dreimaligen Kontakt mit dem Spike-Protein benötigen, damit es nach jetzigem Wissensstand zu einer sehr guten neutralisierenden Aktivität gegen alle VOCs, inklusive Omikron, kommt.“
Wie die Forschenden berichten, sind hier unterschiedliche Konstellationen möglich. Dreifach geimpfte Personen ohne vorige SARS-CoV-2-Infektion kamen fast auf gleiche Titer neutralisierender Antikörper gegen Omikron wie geimpfte Genesene oder Personen, die eine Durchbruchsinfektion mit dem Delta oder Omikron hatten. Prof. Keppler: „In allen Fällen erreichte die Neutralisationsaktivität in unserer Analyse ähnlich hohe Bereiche, und die Bindungsstärke der Antikörper hat sich in allen Konstellationen erhöht.“ Prof. Protzer und Prof. Knolle sind sich einig: „Die durch eine Impfung aufgebaute beziehungsweise verstärkte Immunität ist der Schlüssel zu einem effektiven Schutz vor zukünftigen Varianten des Virus. Aber auch eine Durchbruchsinfektion, so ärgerlich sie ist, erreicht den Effekt einer zusätzlichen Impfung.“
Original Publikation:
Three exposures to the spike protein of SARS-CoV-2 by either infection or vaccination elicit superior neutralizing immunity to all variants of concern.
Wratil PR, Stern M, Priller A, Willmann A, Almanzar G, Vogel E, Feuerherd M, Cheng CC, Yazici S, Christa C, Jeske S, Lupoli G, Vogt T, Albanese M, Mejías-Pérez E, Bauernfried S, Graf N, Mijocevic H, Vu M, Tinnefeld K, Wettengel J, Hoffmann D, Muenchhoff M, Daechert C, Mairhofer H, Krebs S, Fingerle V, Graf A, Steininger P, Blum H, Hornung V, Liebl B, Überla K, Prelog M, Knolle P, Keppler OT, Protzer U.
Nat Med. 2022 Jan 28. doi: 10.1038/s41591-022-01715-4.
Dynamics of spike-and nucleocapsid specific immunity during long-term follow-up and vaccination of SARS-CoV-2 convalescents.
Koerber N, Priller A, Yazici S, Bauer T, Cheng CC, Mijočević H, Wintersteller H, Jeske S, Vogel E, Feuerherd M, Tinnefeld K, Winter C, Ruland J, Gerhard M, Haller B, Christa C, Zelger O, Roggendorf H, Halle M, Erber J, Lingor P, Keppler O, Zehn D, Protzer U, Knolle PA.
Nat Commun. 2022 Jan 10;13(1):153. doi: 10.1038/s41467-021-27649-y.
Abwasser-Monitoring als Pandemie-Frühwarnsystem für Metropolen
Ein Forschungsteam unter der Leitung des Tropeninstituts am LMU Klinikum München hat die Ergebnisse einer der ersten und bisher längsten Untersuchungen zur Abwasserüberwachung auf das neuartige Coronavirus in Bayern in der Fachzeitschrift Science of the Total Environment veröffentlicht.
Die einjährige Verlaufsstudie im Münchner Stadtgebiet bestätigt das Potential des Abwasser-Monitorings, um die Dynamik der Pandemie und die Ausbreitung neuer Virusvarianten frühzeitig zu erkennen.
Lokale Abwasseruntersuchungen erfolgen seit dem Auftreten der Pandemie ab 2020. Im Frühjahr 2021 bekräftigte die Europäische Kommission in ihrer Empfehlung, eine systematische Abwasser-Überwachung von SARS-CoV-2 und seinen Varianten in allen EU-Mitgliedstaaten einzuführen. Besonders in Metropolen, in denen sich das Virus rapide verbreiten kann, bietet die Abwasser-basierte Epidemiologie (Waste-water based epidemiology, WBE) große Vorteile.
Die einjährige Verlaufsstudie, die im April 2020 startete, ist eine der ersten und längsten Untersuchungen zur Nachverfolgung der SARS-CoV-2 RNA Viruslast im Abwasser weltweit und insbesondere in Deutschland. Nun wurden die Ergebnisse in der Fachzeitschrift Science of the Total Environment veröffentlicht. Unter der Leitung des Tropeninstituts am LMU Klinikum München beteiligten sich die Virologie des Max von Pettenkofer-Instituts, das Genzentrum der LMU, die Münchner Stadtentwässerung, die Branddirektion München, das Gesundheitsreferat der Stadt München und die Task Force Infektiologie des Bayerischen Landesamts für Gesundheit und Lebensmittelsicherheit (LGL) an der Studie.
Finanziert wurde die Forschung durch das Bayerische Ministerium für Wissenschaft und Kunst. Wissenschaftsminister Bernd Sibler betont die praktische Relevanz der Untersuchung: „Sie leistet einen wertvollen Betrag, trotz der anhaltenden Corona-Pandemie weitere Schritte zur Normalisierung des öffentlichen Lebens in den Blick zu nehmen. Deshalb haben wir die Studie Prospektive COVID-19 Kohorte München - KoCo19 mit rund einer Million Euro unterstützt.“
In ihrer Untersuchung im Raum München sammelten die Wissenschaftlerinnen und Wissenschaftler seit Anfang April 2020 über ein Jahr lang wöchentlich Abwasserproben an sechs Standorten im Münchner Stadtgebiet. Die Proben wurden im Labor mittels RT-PCR (reverse transcription polymerase chain reaction) und Genomsequenzierung untersucht, um die Verbreitung von SARS-CoV-2 in München im zeitlichen und räumlichen Verlauf zu messen. Durch die Entschlüsselung des Erbguts anhand der Sequenzierung isolierter Virus-RNA erhielten die Forscher zudem Informationen, ob in den Proben besorgniserregende Virusvarianten (variants of concern, VOC) auftraten.
Die Ergebnisse zeigten, dass die in den Proben nachgewiesene SARS-CoV-2-RNA-Last gut mit den offiziellen Daten der 7-Tage-Inzidenz in den jeweiligen Stadtgebieten übereinstimmte. Studienleiter PD Dr. med. Andreas Wieser vom Tropeninstitut am LMU Klinikum berichtet: “Durch die im Abwasser gemessene Viruslast haben wir die lokale Inzidenz für die Verbreitung von SARS-CoV-2 bereits drei Wochen früher festgestellt als in den Meldezahlen der Behörden, die auf der Analyse von Atemwegsabstrichen basieren. Zudem konnten wir die zunehmende Ausbreitung der Virusvariante B.1.1.7 (Alpha) in der Münchner Bevölkerung bereits Anfang Januar 2021 nachweisen, Wochen bevor diese durch die Sequenzierung von Abstrich-Proben von Patienten in München in relevanter Zahl festgestellt werden konnte.”
Insgesamt bestätigt die Studie das Potential des Abwasser-Monitorings als Frühwarnsystem für die SARS-CoV-2 Pandemie und die Vorteile im Vergleich zu Massentests durch Abstriche. Dies gilt insbesondere hinsichtlich der Verbreitung besorgniserregender Virusvarianten in der lokalen Bevölkerung. Außerdem weist die Abwasser-Beprobung weniger Verzerrungen z.B. durch geänderte Regeln bei der Probenahme oder bei Meldewegen auf, als dies bei auf Abstrich-/Meldedaten basierten Statistiken der Fall ist.
Original Publikation:
Spatially resolved qualified sewage spot sampling to track SARS-CoV-2 dynamics in Munich - One year of experience.
Rubio-Acero R, Beyerl J, Muenchhoff M, Roth MS, Castelletti N, Paunovic I, Radon K, Springer B, Nagel C, Boehm B, Böhmer MM, Graf A, Blum H, Krebs S, Keppler OT, Osterman A, Khan ZN, Hoelscher M, Wieser A; KoCo19-Study Group.
Sci Total Environ. 2021 Jul 21;797:149031. doi: 10.1016/j.scitotenv.2021.149031.
LMU und PEI Forscher entwickeln effektiven und sicheren RNA Replikon Impfstoff gegen COVID-19
Die anhaltende Covid-19 Pandemie kann mit nur mit effektiven und sicheren Impfstoffen unter Kontrolle gebracht werden. Während die aktuellen mRNA Impfstoffe von BioNTech/Pfizer und Moderna als sicher gelten, bestehen bei einigen DNA Virusvektor-Impfstoffen bisher Bedenken oder Befangenheiten hinsichtlich ihrer Unschädlichkeit oder unzureichenden Wirksamkeit. Wissenschaftler der Ludwig-Maximilians-Universität (LMU) um Alexandru Hennrich und Karl-Klaus Conzelmann am Max von Pettenkofer-Institut der LMU und Genzentrum und des Paul-Ehrlich-Instituts (PEI) um Christian Pfaller, Forschungseinheit Pathogenese von Atemwegsviren, haben einen im Tiermodell hochwirksamen und sicheren RNA Replikon Impfstoff entwickelt.
Der neuartige Impfstoff besteht aus einer selbst-replizierenden RNA, einem sogenannten Replikon, das aus einem Tier-Rhabdovirus, dem Virus der vesikulären Stomatitis (VSV), abgeleitet ist. Das Replikon kodiert den Teil des Coronavirus Spike Proteins, der für die Ausbildung einer schützenden Immunität verantwortlich ist. Im Labor kann das Replikon in eine VSV Lipid-Hülle verpackt werden, um die RNA bei der Impfung in Muskel- und Immunzellen zu schleusen, und dort das schützende Spike Antigen zu produzieren.
Besondere Sorgfalt wurde auf die Konstruktion und Präsentation des Antigens verwendet. Während in den bisherigen Impfstoffen komplettes Coronavirus Spike Protein eingesetzt wird, kodiert das VSV Replikon ein chimäres, membranständiges „Minispike“, auf dessen Spitze lediglich die Rezeptor-bindende Domäne (RBD) des Coronavirus Spike präsentiert wird. Wie inzwischen bekannt ist, sind die gegen COVID-19 schützenden Virus-neutralisierenden Antikörper fast ausschließlich gegen die RBD gerichtet. Die Produktion von unwirksamen oder potentiell schädlichen Antikörpern durch das Immunsystem wird dadurch vermieden. Das chimäre Antigen wird nach der Infektion durch den Vektorimpfstoff auf der Zelloberfläche präsentiert und regt das Immunsystem zur Bildung Antikörpern an, die die Infektion durch das natürliche SARS-CoV-2 Coronavirus blockieren.
Die Besonderheit des Minispikes liegt darin, dass der membranständige Teil einem Rhabdovirus Glykoprotein nachempfunden ist, welches die Replikon-RNA in den infizierten Zellen wieder mobilisieren kann. So werden nichtinfektiöse Virus-ähnliche Partikel (virus-like-particles; VLPs) freigesetzt, die das Minispike an ihrer Oberfläche tragen und das Immunsystem nochmals weiter stimulieren. Das System stellt damit eine Kombination aus zwei unterschiedlichen Impfstoffen, einem konventionellen Vektorimpfstoff und einem VLP Impfstoff dar, den die Autoren als 2-in-1 Ansatz bezeichnen. Im Tierexperiment wurde gezeigt, dass nach einer 2-in-1 Impfung sehr hohe Antikörper Titer produziert werden und dass transgene, für SARS-CoV-2 permissive Mäuse durch eine einzige Immunisierung vor der COVID-19 Erkrankung geschützt werden. Die Notwendigkeit einer Nach-Impfung (Boost-Immunisierung) wie sie bei einfachen Impfstoffen oft notwendig ist, entfällt somit.
Die Autoren sehen in dem neuen VSV-Minispike Impfstoff Ansatz eine geeignete Alternative für die COVID-19 Impfung von Personen mit geschwächtem Immunsystem und einen vielversprechenden Ausgangspunkt für die Entwicklung von Impfstoffen für Pathogene, gegen die bisher noch nicht erfolgreich geimpft werden kann. Die Befunde wurden in der Fachzeitschrift PLoS Pathogens veröffentlicht:
Safe and effective two-in-one replicon-and-VLP minispike vaccine for COVID-19: Protection of mice after a single immunization
Hennrich AA, Sawatsky B, Santos-Mandujano R, Banda DH, Oberhuber M, Schopf A, Pfaffinger V, Wittwer K, Riedel C, Pfaller CK, Conzelmann KK.
PLoS Pathog. 2021 Apr 21;17(4):e1009064
COVID-19: Zellen im Shutdown
Strukturbiologen der LMU zeigen im Detail, wie das neuartige Coronavirus die Proteinproduktion an den Ribosomen lahmlegt und wichtige Teile der angeborenen Immunabwehr ausschaltet.
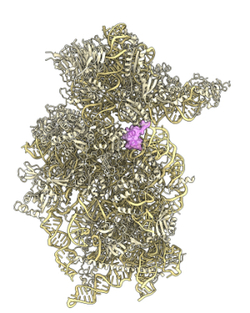 Der Name und das Kürzel des Viruseiweißes klingen unauffällig, doch seine Wirkung ist verheerend: Das Nonstructural Protein 1 (Nsp1) ist eine der zentralen Waffen, die das neuartige Coronavirus SARS-CoV-2 einsetzt, um sich im menschlichen Körper auszubreiten. Bekannt war dieser Virulenzfaktor seit dem Ausbruch des verwandten SARS-Coronavirus vor gut zehn Jahren: Nsp1, so fanden Wissenschaftler bereits damals heraus, legt im Wirt die Proteinproduktion an den Ribosomen lahm. Doch nun haben Forscher der LMU und des Universitätsklinikums Ulm herausgefunden, was Nsp1 so gefährlich macht, und beschreiben detailliert die Mechanismen des Angriffs. Die Arbeit ist am 17. Juli im renommierten Wissenschaftsmagazin Science erschienen.
Der Name und das Kürzel des Viruseiweißes klingen unauffällig, doch seine Wirkung ist verheerend: Das Nonstructural Protein 1 (Nsp1) ist eine der zentralen Waffen, die das neuartige Coronavirus SARS-CoV-2 einsetzt, um sich im menschlichen Körper auszubreiten. Bekannt war dieser Virulenzfaktor seit dem Ausbruch des verwandten SARS-Coronavirus vor gut zehn Jahren: Nsp1, so fanden Wissenschaftler bereits damals heraus, legt im Wirt die Proteinproduktion an den Ribosomen lahm. Doch nun haben Forscher der LMU und des Universitätsklinikums Ulm herausgefunden, was Nsp1 so gefährlich macht, und beschreiben detailliert die Mechanismen des Angriffs. Die Arbeit ist am 17. Juli im renommierten Wissenschaftsmagazin Science erschienen.
Ein wichtiger Virulenzfaktor von SARS-CoVs ist das Nichtstrukturprotein 1 (Nsp1). Seit dem Ausbruch der verwandten SARS-CoV vor mehr als 10 Jahren ist bekannt, dass Nsp1 die Produktion von Wirtsproteinen unterdrücken kann, indem es durch einen ungeklärten Mechanismus mit der ribosomalen 40S-Untereinheit assoziiert. Die kleinen 40S- und die großen 60S-Ribosomen-Untereinheiten bilden zusammen die zelluläre Maschinerie zur Proteinproduktion, das 80S-Ribosom, das unter Verwendung der mRNA als Vorlage Proteine produziert (Translation).

Die Beckmann-Gruppe beschäftigt sich seit Jahrzehnten mit Ribosomen und Translation und hat zusammen mit der Sparrer-Gruppe der Universität Ulm beschlossen, Nsp1 aus dem aktuellen Coronavirus genauer zu analysieren. Sie fanden heraus, dass auch das Nsp1-Protein aus dem pandemischen SARS-CoV-2 an 40S-Untereinheiten aber auch an 80S-Ribosomen bindet. Infolgedessen wird die Übersetzung der zellulären mRNA in Proteine sowohl im in vitro Experiment als auch in menschlichen Zellen fast vollständig abgeschaltet. Die so inaktivierten Ribosomen konnten bis ins molekulare Detail mittels Kryo-Elektronenmikroskopie bei einer Auflösung von 2,6 Å sichtbar gemacht werden, sodass erkennbar war wie Nsp1 in den Tunnel für die mRNA bindet und dadurch das Ribosom inaktiviert. Dies war nur möglich mit Hilfe der in den letzten Jahren etablierten High-End-Ausrüstung der Kryo-Elektronenmikroskopie Einrichtung des Genzentrums, die gemeinsam von der LMU und der Deutschen Forschungsgemeinschaft (DFG) finanziert wurde.
Interessanterweise hat die Sparrer-Gruppe von der Universität Ulm in menschlichen Zellen beobachtet, dass die weitgehende Abschaltung der Translation durch Nsp1 eine fast vollständige Hemmung der angeborenen Immunantwort zur Folge hat. Dies ist eines der wichtigsten zellulären Abwehrsysteme gegen Viren, welches somit durch Nsp1 neutralisiert wird und dadurch zur Immunevasion des Virus beiträgt. Daher könnte die Entschlüsselung des molekularen Hemmmechanismus von Nsp1 als Ausgangspunkt für die Entwicklung strukturbasierter Medikamente gegen SARS-CoV-2 dienen.
Animierte Zusammenfassung der Forschungsergebnisse
Bericht im BR Fernsehen vom 01.08.2020: Corona-Virus und Immunsystem
Auf der ganzen Welt forschen Wissenschaftler, um die Ausbreitung des Corona-Virus zu bekämpfen. Manchmal hat man den Eindruck, sie arbeiteten nicht so eng zusammen, wie es eigentlich nötig wäre. Aber es gibt auch Positivbeispiele! In München und Ulm machen Wissenschaftler gemeinsame Sache und haben rausgefunden, wie Corona unser Immunsystem lahmlegen kann.
Zur Pressemeldung der LMU
Zur Pressemeldung der Universität Ulm
Mehr zur Forschung von Prof. Roland Beckmann
Vielversprechender therapeutischer Ansatz bei SARS-CoV-2 Infektion
Die Arbeitsgruppe von Frau Prof. Marion Subklewe untersucht die adaptive (erworbene) Immunantwort bei Patienten, die an einer SARS-CoV-2 Infektion leiden. Im Vordergrund stehen dabei die T-Zellen, eine Gruppe von weißen Blutzellen, die der Immunabwehr dienen. Diese werden hinsichtlich auftretender Unterarten und verschiedener Expressionsmarker charakterisiert. Der Wirkstoff Tocilizumab, ein IL-6 Rezeptor Antikörper, stellt einen vielversprechenden therapeutischen Ansatz der SARS-CoV-2 Infektion dar. Der immunmodulatorische Einfluss dieser Substanz auf das adaptive Immunsystem wird ebenfalls untersucht. Die Medizinische Klinik III des Klinikums der LMU ist das erste Prüfzentrum in Deutschland, welches die Wirksamkeit von Tocilizumab bei SARS-CoV-2 Infektionen in einer randomisierten Placebo-kontrollierten Studie prüft.
Mehr zur Forschung von Prof. Marion Subklewe
Pressemeldung LMU Klinikum vom 14.04.2020
Smartphone-App ermöglicht engmaschige digitale Nachsorge von Covid-19-Patienten
Die Ärzte des LMU Klinikums nutzen das COVID-19 Caregiver Cockpit von Cankado zur medizinischen Überwachung ihrer entlassenen Covid-19-Patienten, um so mögliche Komplikationen rechtzeitig zu erkennen - und gegebenenfalls zeitnah reagieren zu können. Ideengeberin war Prof. Marion Subklewe: Die Oberärztin der Medizinischen Klinik und Poliklinik III des LMU Klinikums setzt die App schon seit Längerem für die Betreuung von Krebspatienten ein und erkannte, dass die Anwendungssoftware auch für die digitale Nachsorge von Covid-19-Patienten genutzt werden kann.
Pressemeldung LMU Klinikum vom 04.05.2020
Herstellung und Nachweisverfahren für SARS-CoV-2 Antikörper
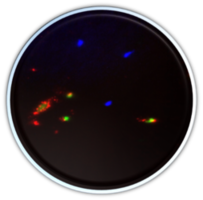 Die Arbeitsgruppe von Prof. Karl-Klaus Conzelmann entwickelt auf der Basis von selbst-replizierenden RNAs SARS-CoV-2 Antigen Präsentationssysteme für Immunisierungszwecke und serologische Untersuchungen. In Zusammenarbeit mit dem Helmholtz-Zentrum München (HMGU) werden monoklonale Antikörper hergestellt, die für Antigen-Tests während der akuten Infektion eingesetzt werden sollen und die bisher benutzten Nukleinsäure-tests (RT-PCR) ergänzen können. Ein Immunfluoreszenztest mit SARS-1 und SARS-2 spezifischem Antigen wird für den Nachweis von COVID19-spezifischen Antikörpern (in der Abb. rot) bei erkrankten und genesenen Patienten eingesetzt. Solche serologischen Tests werden auch zur Bestimmung der Durchseuchungsrate benötigt.
Die Arbeitsgruppe von Prof. Karl-Klaus Conzelmann entwickelt auf der Basis von selbst-replizierenden RNAs SARS-CoV-2 Antigen Präsentationssysteme für Immunisierungszwecke und serologische Untersuchungen. In Zusammenarbeit mit dem Helmholtz-Zentrum München (HMGU) werden monoklonale Antikörper hergestellt, die für Antigen-Tests während der akuten Infektion eingesetzt werden sollen und die bisher benutzten Nukleinsäure-tests (RT-PCR) ergänzen können. Ein Immunfluoreszenztest mit SARS-1 und SARS-2 spezifischem Antigen wird für den Nachweis von COVID19-spezifischen Antikörpern (in der Abb. rot) bei erkrankten und genesenen Patienten eingesetzt. Solche serologischen Tests werden auch zur Bestimmung der Durchseuchungsrate benötigt.
Mehr zur Forschung von Prof. Karl-Klaus Conzelmann
Optimierung von Nachweisverfahren für SARS-CoV-2 Antikörper
Eine Infektion mit SARS-CoV-2 führt zu einer Immunreaktion, bei der spezifische Antikörper gebildet werden. Diese sind im Blut von erkrankten und genesenen Personen nachweisbar. Deshalb besteht ein enormer weltweiter Bedarf an sensitiven und spezifischen serologischen Nachweisen von SARS-CoV-2 Antikörpern. Die Herausforderung besteht darin, empfindliche Tests zu entwickeln, die jedoch nicht auf z.B. andere Coronaviren (z.B. Erkältungsviren) ansprechen. Die Arbeitsgruppe Hopfner stellt rekombinantes Protein aus SARS-CoV-2 her, um in Zusammenarbeit mit den Arbeitsgruppen Immunbiochemie (Prof. Hornung) und „Monoclonal Antibodies“ (Dr. Kremmer und Dr. Flaswinkel, Lehrstuhl Prof Leonhardt) Verfahren und Reagenzien für ELISA basierte Nachweismethoden zu optimieren.
Mehr zur Forschung von Prof. Karl-Peter Hopfner
Sequenzierung von SARS-CoV-2 Genomen
 Während der Ausbreitung verändert sich die genetische Information von SARS-Cov-2 ständig. Durch die Erfassung dieser Veränderung können wertvolle Erkenntnisse über Ausbreitungswege, Infektionsketten und Evolution von SARS-CoV-2 gewonnen werden. Die Arbeitsgruppen von Dr. Helmut Blum und von Prof. Oliver T. Keppler haben gemeinsam ein schnelles und kostengünstiges Verfahren zur Sequenzierung des SARS-CoV-2 Genoms etabliert. Aus Rachenabstrichen von Patienten wird das Virusgenom zunächst in einer Multiplex-PCR vermehrt und dann mit der Nanoporen-Sequenzierung bestimmt. Die Ergebnisse fließen in klinische und epidemiologische Studien ein und werden der wissenschaftlichen Gemeinschaft über die frei zugängliche Plattformen GISAID und Nextstrain zur Verfügung gestellt.
Während der Ausbreitung verändert sich die genetische Information von SARS-Cov-2 ständig. Durch die Erfassung dieser Veränderung können wertvolle Erkenntnisse über Ausbreitungswege, Infektionsketten und Evolution von SARS-CoV-2 gewonnen werden. Die Arbeitsgruppen von Dr. Helmut Blum und von Prof. Oliver T. Keppler haben gemeinsam ein schnelles und kostengünstiges Verfahren zur Sequenzierung des SARS-CoV-2 Genoms etabliert. Aus Rachenabstrichen von Patienten wird das Virusgenom zunächst in einer Multiplex-PCR vermehrt und dann mit der Nanoporen-Sequenzierung bestimmt. Die Ergebnisse fließen in klinische und epidemiologische Studien ein und werden der wissenschaftlichen Gemeinschaft über die frei zugängliche Plattformen GISAID und Nextstrain zur Verfügung gestellt.
Mehr zur Forschung von Dr. Helmut Blum
Hochdurchsatz-Verfahren für die SARS-CoV-2 Diagnostik
Zu Beginn der Pandemie bestand bei vielen Laboren ein Engpass in der SARS-CoV-2-PCR-Diagnostik: die Extraktion von viralen Nukleinsäuren aus Nasen-Rachen-Abstrichen und anderem Patientenmaterial der oberen Atemwege. Die viralen Nukleinsäuren werden für den anschließende PCR-Analyseschritt benötigt. Um den steigenden Anforderungen gerecht zu werden, entwickelte der Automatisierungsspezialist Jochen Rech aus dem Labor Hornung in kurzer Zeit eine Hochdurchsatz-Nukleinsäure-Extraktionsmethode. Diese ermöglicht die gleichzeitige Bearbeitung von 96 Proben in 30 Minuten. Mit diesem Aufbau konnten die Testkapazitäten des Max-von-Pettenkofer-Instituts, des Genzentrums der LMU und seines Universitätsklinikums (Leiter der Virologie: Prof. Oliver T. Keppler) um bis zu 800 Proben pro Tag erhöht werden.
Die Untersuchungen werden im Rahmen von SARS-CoV-2-Forschungsstudien durchgeführt und auch in die qualitätskontrollierte Routinediagnostik von klinischen Proben implementiert.
Mehr zur Forschung von Prof. Veit Hornung
Medienbeiträge mit Virologe Prof. Oliver T. Keppler
Oliver T. Keppler, Professor für Virologie am Max-von-Pettenkofer-Institut und Mitglied des Genzentrums, ist ein gefragter Experte bei der Coronavirus Pandemie.
Alle Beträge mit Prof. Keppler auf BR24 finden Sie hier.
Three exposures to spike protein for a broad immunity
Since the beginning of the Covid-19 pandemic, SARS-CoV-2 has continued to evolve, with new variants of concern (VoCs) spreading rapidly. Highly contagious and partially capable of evading the immune response, Omicron has become the dominant variant in most countries. Scientists from LMU, Helmholtz Munich, and TUM have shown that the immune system is capable of neutralizing even Omicron after a total of three exposures to the viral spike protein.
Sewage monitoring as a pandemic early warning system for cities
A research team now confirms the great potential of sewage monitoring as an early warning system for metropolitan areas, using the city of Munich as an example. The team led by the Tropical Institute at LMU Klinikum München has published the results of one of the first and so far longest sewage monitoring studies for the novel coronavirus in Bavaria in the journal Science of the Total Environment.
The scientists collected weekly wastewater samples at six locations in the Munich city area for over a year from the beginning of April 2020. The samples were analysed in the laboratory using RT-PCR (Reverse Transcription Polymerase Chain Reaction) and genome sequencing to measure the spread of SARS-CoV-2 in Munich over time and space. By decoding the genetic material by sequencing isolated viral RNA, the researchers also obtained information on whether variants of concern (VOC) occurred in the samples.
Researchers at LMU and PEI develop safe and effective RNA replicon vaccine against COVID-19
Sustainable control of the current COVID-19 pandemic requires effective and safe vaccines. While the front-runner mRNA vaccines from BioNTech/Pfizer and Moderna seem to be safe, concerns exist with a number of virus-vectored vaccines in terms of safety or efficacy. Scientists around Alexandru Hennrich and Karl-Klaus Conzelmann (Max von Pettenkofer Institute and Gene Center of Ludwig-Maximilians-Universität) (LMU), and Christian Pfaller (Research unit pathogenesis of airway viruses at the Paul-Ehrlich-Institut (PEI), have joint to develop an RNA replicon vaccine which is highly efficient in animal models.
Visualization of the SARS-CoV-2 virulence factor Nsp1 hijacking human ribosomes
 The severe acute respiratory syndrome-coronavirus 2 (SARS-CoV-2), the cause of the COVID-19 pandemic, has infected millions worldwide with a death toll amounting to hundreds of thousands. Neither vaccines nor efficient drugs are available so far. Researchers from LMU Gene Center and university of Ulm have determined how the pandemic coronavirus SARS-CoV-2 inhibits the synthesis of proteins in infected cells and shown that it effectively disarms the body’s innate immune system.
The severe acute respiratory syndrome-coronavirus 2 (SARS-CoV-2), the cause of the COVID-19 pandemic, has infected millions worldwide with a death toll amounting to hundreds of thousands. Neither vaccines nor efficient drugs are available so far. Researchers from LMU Gene Center and university of Ulm have determined how the pandemic coronavirus SARS-CoV-2 inhibits the synthesis of proteins in infected cells and shown that it effectively disarms the body’s innate immune system.
Although its name is relatively unspecific and indeed opaque, the Nonstructural Protein 1 (Nsp1) encoded by the coronavirus SARS-Cov-2, which is responsible for the current pandemic, has now been shown to have a devastating effect on host cells. Nsp1 is in fact one of the central weapons used by the virus to ensure its own replication and propagation in human hosts. Nsp1 was identified as a virulence factor following the outbreak of the related SARS coronavirus nearly 20 years ago, when it was shown to inhibit protein synthesis in infected cells. Now researchers based at LMU and Ulm University Hospital have discovered what makes Nsp1 so potent. In a paper which appears in the journal Science, they describe the protein’s mode of action in detail.

The Beckmann group has studied ribosomes and translation for decades and, together with the Sparrer group of Ulm University, decided to analyze Nsp1 from the current Coronavirus in more detail. They found that the Nsp1 protein from pandemic SARS-CoV-2 binds to both, 40S subunits and to 80S ribosomes. As a result, translation of cellular mRNA is shutdown both, in vitro and in human cells. By high-end cryo-electron microscopy (cryo-EM) they discovered that the C-terminus of Nsp1 inserts into the mRNA entry channel as a hairpin of alpha-helices and blocks it for mRNA, thus explaining translation inhibition by Nsp1. Notably, binding in this channel depended strictly on two specific amino acid side chains of Nsp1, which were inserted into a distinct pocket on the ribosome. These important molecular details were resolved at a resolution of 2.6 Å, which was only possible with the recently established high-end equipment of the Gene Center cryo-EM facility, jointly funded by the LMU and the Deutsche Forschungsgemeinschaft (DFG). Moreover, a cryo-EM inventory of native Nsp1-ribosome complexes, which were purified from transfected human cells, revealed 40S and unusual 80S ribosomes, all displaying the same mode of Nsp1 blocking the mRNA entry channel. Also in human cells, the Sparrer group of Ulm University observed that shutdown of translation by Nsp1 is responsible for a near complete inhibition of the innate immune response, one of the major cellular virus defense systems, thereby contributing to the immune evasion strategy of the virus. Thus, having deciphered in detail the molecular basis of the inhibitory mechanism of Nsp1 may serve as starting point for structure-based drug design against SARS-CoV-2.
LMU press release
University of Ulm press release
Promising therapeutic approach for SARS-CoV-2 infection
The research group of Prof. Marion Subklewe investigates the adaptive immune response in patients suffering from SARS-CoV-2 infection. In particular, the T cells are characterized with respect to subtypes and different expression markers. Tocilizumab, an IL-6 receptor antibody, represents a promising therapeutic approach for SARS-CoV-2 infection. The immunomodulatory influence of this substance on the adaptive immune system is also investigated. The Medical Clinic III of the LMU hospital is the first test center in Germany to evaluate the efficacy of tocilizumab in a randomized placebo-controlled study.
Smartphone app enables close digital follow-up of Covid-19 patients
The doctors at the LMU Klinikum use Cankado's COVID-19 Caregiver Cockpit for the medical monitoring of their discharged Covid-19 patients in order to detect possible complications in time - and to react promptly if necessary. The idea was inspired by Prof. Marion Subklewe: The senior physician of the Medical Clinic and Polyclinic III of the LMU Hospital has been using the app for a long time for the care of cancer patients and recognized that the application software can also be used for the digital follow-up care of Covid-19 patients.
Development and detection methods for SARS-CoV-2 antibodies
 The research group of Prof. Conzelmann develops presentation systems for SARS-CoV-2 antigen based on self-replicating RNAs for immunization and serological investigations. In collaboration with Helmholtz Munich (HMGU) monoclonal antibodies (MAbs) are generated which shall be used for detection of virus in acute infections and which could complement the widely used PCR tests. An immunofluorescence test using SARS-1 and SARS-2 specific antigens is employed for demonstration of COVID-19 specific antibodies of diseased and recovered patients and useful for determining infection rates.
The research group of Prof. Conzelmann develops presentation systems for SARS-CoV-2 antigen based on self-replicating RNAs for immunization and serological investigations. In collaboration with Helmholtz Munich (HMGU) monoclonal antibodies (MAbs) are generated which shall be used for detection of virus in acute infections and which could complement the widely used PCR tests. An immunofluorescence test using SARS-1 and SARS-2 specific antigens is employed for demonstration of COVID-19 specific antibodies of diseased and recovered patients and useful for determining infection rates.
Optimization of detection methods for SARS-CoV-2 antibodies
Sensitive and specific serological detection of SARS-CoV-2 in sick and recovered patients is a worldwide need in the context of COVID-19. The tests are supposed to be highly sensitive but should not detect e.g. corona viruses that cause the common cold. The Hopfner lab is producing recombinant protein from SARS-CoV-2 in order to help optimize methods and provide reagents for ELISA procedures together with the groups Immunobiochemistry (Prof. Hornung) and “Monoclonal Antibodies” (Dr. Kremmer and Dr. Flaswinkel, Chair Prof. Leonhardt).
Sequencing of SARS-CoV-2 genomes
 During the spread, the genetic information of SARS-Cov-2 is constantly changing. By recording these changes, valuable insights into the propagation pathways, infection chains and evolution of SARS-CoV-2 can be gained. The research groups of Dr. H. Blum and Prof. Oliver T. Keppler have jointly established a fast and cost-effective method for sequencing of SARS-Cov-2 genomes. From throat swabs of patients the virus genome is first amplified in a multiplex PCR and then determined by nanopore sequencing. The results will be used in clinical and epidemiological studies and will be made available to the scientific community via the freely accessible platforms GISAID and Nextstrain.
During the spread, the genetic information of SARS-Cov-2 is constantly changing. By recording these changes, valuable insights into the propagation pathways, infection chains and evolution of SARS-CoV-2 can be gained. The research groups of Dr. H. Blum and Prof. Oliver T. Keppler have jointly established a fast and cost-effective method for sequencing of SARS-Cov-2 genomes. From throat swabs of patients the virus genome is first amplified in a multiplex PCR and then determined by nanopore sequencing. The results will be used in clinical and epidemiological studies and will be made available to the scientific community via the freely accessible platforms GISAID and Nextstrain.
High throughput diagnostics for SARS-CoV-2
At the beginning of the corona pandemic many laboratories faced a bottleneck in SARS-CoV-2 PCR diagnostics: the extraction of viral nucleic acids from nasopharyngeal swabs and other respiratory material for subsequent PCR analysis. To meet these increasing demands, automation specialist Jochen Rech from the Hornung laboratory implemented a high throughput nucleic acid extraction methodology that allows the simultaneous processing of 96 samples in 30 minutes. With this setup at hand, the testing capacities of the Max von Pettenkofer Institute and Gene Center of the LMU and its university hospital (head of virology: Prof. Oliver T. Keppler) could be increased by up to 800 samples per day. Testings will be performed in the context of SARS-CoV-2 research studies and also be implemented into quality-controlled routine diagnostics of clinical samples.

